
|
 kod genetyczny
kod genetyczny
 Cząsteczka DNA jest nośnikiem informacji genetycznej, niezbędnej do reprodukcji organizmu.
Cząsteczka DNA jest nośnikiem informacji genetycznej, niezbędnej do reprodukcji organizmu.Odczytywanie tej informacji z wykorzystaniem w syntezie białek nazywamy ekspresją genów. W procesie tym informacja zawarta w DNA zostaje przepisana na RNA, które następnie kieruje syntezą białka.
Sekwencja zasad na kodującej nici DNA w kierunku 5`→3`, określa kolejność aminokwasów polipeptydu (kierunek od aminowego do karboksylowego).
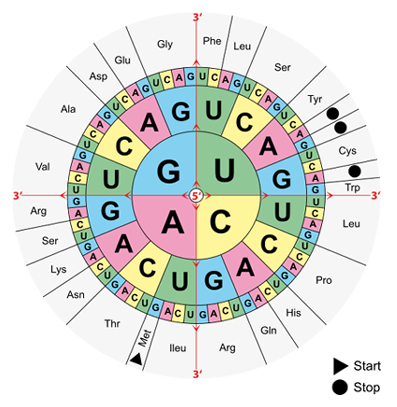
Nie ma tu miejsca na żaden bałagan, czy przypadkowość. Sposób, w jaki sekwencja zasad kwasu nukleinowego zostanie przekształcona w sekwencję aminokwasów podczas biosyntezy białek jest ściśle określony, narzucony i opisany przy pomocy kodu genetycznego, posługującego się kodonami. Kodon z kolei jest to układ trzech zasad tworzących jednostkę (kodon), będącą składową sekwencji DNA genu.
Ponieważ cztery zasady w DNA mogą utworzyć 64 kodony (43) określające 20 aminokwasów występujących w białkach, a zatem liczba kodonów jest większa od liczby białkowych aminokwasów, są więc one kodowane przez więcej niż jeden kodon, co nazywamy degeneracją. Wyjątek tutaj stanowią metionina i tryptofan - kodowane jednym kodonem.
Kodony "obsługujące" te same aminokwasy są podobne (różnią się głównie w trzeciej pozycji - pozycja tolerancji) i nazywane są synonimami, np. kodujące treoninę ACU, ACC, ACA i ACG.
Chociaż degeneracja kojarzy nam się nie koniecznie pozytywnie, to w przypadku kodu genetycznego minimalizuje ona efekty mutacji i ich wpływ na funkcje białek.
Do kodowania aminokwasów wykorzystywane są 61 (spośród 64) kodony. Pozostałe trzy (UAG, UGA, UAA) nie kodują, lecz stanowią sygnał zakończenia translacji i są określane jako kodony terminacyjne lub kodony stop.(co nieco przypomina nadawanie telegramu)
Słusznie ktoś spyta w tym momencie, czy jeśli posiadamy kodony stop, to mamy również kodony start? I owszem.
Kodonem start (lub inaczej inicjującym), który dla wszystkich białek jest sygnałem startu translacji jest kodon metioninowy AUG.
Kodon inicjujący spełnia kilka funkcji: rozpoczyna wszystkie polipeptydy, jak również określa ramkę odczytu RNA.
Z każdej sekwencji RNA można odczytać trzy zestawy kodonów, z czego tylko jedna ramka odczytu zawiera właściwe informacje. Dwie pozostałe zawierają najczęściej kilka kodów stop, co wyklucza możliwość wykorzystania ich w kierowaniu syntezą białka.
Kodony składając się obok siebie tworzą zestawy. Taki wielokodonowy zestaw, zaczynający się kodonem inicjującym i zakończony kodonem terminacyjnym (niczym wieloetapowa trasa ze startem i metą) jest określany jako ORF, czylo Otwarta Ramka Odczytu. ORF wykorzystuje się do identyfikacji sekwencji DNA kodujących białko.
Wiadomo jest, że (choć rzadko spotykane) istnieją różne warianty kodu. Przykładem mogą być tu mitochondria, które zawierają mały genom DRN (ok. 20 genów ze zmianami w stosunku do kodu uniwersalnego). Zmiany dotyczą najczęściej kodonów start i stop. np. UGA (stop) w mitochondriach koduje tryptofan, a zamiast niego funkcję kodonu terminacyjnego przejęły AGA i AGG (normalnie kodujące argininę).
pierwsza pozycja |
pozycja druga |
pozycja trzecie |
|||||||
|---|---|---|---|---|---|---|---|---|---|
U |
C |
A |
G |
||||||
| U | Phe Phe Leu Leu |
UUU UUC UUA UUG |
Ser Ser Ser Ser |
UCU UCC UCA UCG |
Tyr Tyr Stop Stop | UAU UAC UAA UAG |
Cys Cys Stop Trp |
UGU UGC UGA UGG |
U C A G |
| C | Leu Leu Leu Leu |
CUU CUC CUA CUG |
Pro Pro Pro Pro |
CCU CCC CCA CCG |
His His Gln Gln |
CAU CAC CAA CAG |
Arg Arg Arg Arg |
AGU AGC AGA AGG |
U C A G |
| A | Ile Ile Ile Met |
AUU AUC AUA AUG |
Thr Thr Thr Thr |
ACU ACC ACA ACG |
Asn Asn Lys Lys |
AAU AAC AAA AAG |
Ser Ser Arg Arg |
AGU AGC AGA AGG |
U C A G |
| G | Val Val Val Val |
GUU GUC GUA GUG |
Ala Ala Ala Ala |
GCU GCC GCA GCG |
Asp Asp Glu Glu |
GAU GAC GAA GAG |
Gly Gly Gly Gly |
GGU GGC GGA GGG |
U C A G |
Tweet